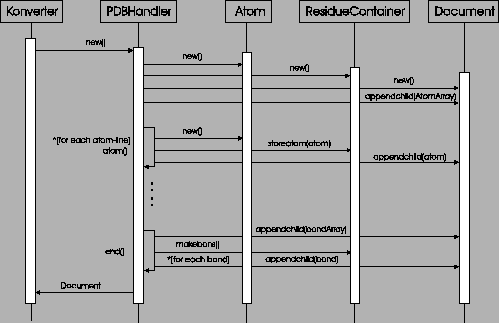
|
DocumentBuilderFactory factory =
DocumentBuilderFactory.newInstance();
DocumentBuilder builder = null;
try {
builder = factory.newDocumentBuilder();
} catch (ParserConfigurationException e){
System.err.println ("ParserConfigurationException "
+ e.getMessage());
System.exit (1);
}
DOMImplementation domIm = builder.getDOMImplementation();
try {
DocumentType docType = domIm.createDocumentType
("molecule", "http://www.xml-cml.org/dtd/cml1_0_1.dtd",
"file:./cml.dtd");
document = domIm.createDocument("cml", "molecule", docType);
Element moleculeNode = (Element) document.getLastChild();
Element atomArray = document.createElement("atomArray");
atomArray.setAttribute("id", "Model 1");
moleculeNode.appendChild(atomArray);
} catch (DOMException e) {
System.err.println ("DOMException " + e.getMessage());
System.exit (1);
}
|
public void atom(String line) {
// auslesen der Zeileninformationen
String atomId = line.substring(6, 11);
String atomName = line.substring(12, 16);
String resName = line.substring(17, 20);
String chainId = line.substring(21, 22);
String resId = line.substring(22, 26);
String xString = line.substring(30, 38).trim();
String yString = line.substring(38, 46).trim();
String zString = line.substring(46, 54).trim();
String occupancy = line.substring(54, 60).trim();
// ermittelt das Elementsymbol
String symbol = atomName.substring(0, 2).trim();
if (Character.isDigit(symbol.charAt(0))) symbol = "H";
// eindeutiger String mit zusaetzlichen Informationen
String id = atomId +":"+ atomName +":"+ resId +
":"+ resName +":"+ chainId;
Element atomNode = document.createElement("atom");
atomNode.setAttribute("title", atomName);
atomNode.setAttribute("id", id);
makeNode("string","builtin","elementType",symbol,atomNode);
makeNode("float","builtin","x3",xString, atomNode);
makeNode("float","builtin","y3",yString, atomNode);
makeNode("float","builtin","z3",zString, atomNode);
makeNode("float","builtin","occupancy",occupancy,atomNode);
Element atomArray =(Element)
((Element) document.getLastChild()).getLastChild();
atomArray.appendChild(atomNode);
Atom atom = null; // Container fuer die Atomdaten
try { atom = new Atom(pse.getElement(symbol)); }
catch (NoSuchElementException e) {
System.err.println("Kein Element "+symbol+" gefunden!");
System.exit(1);
}
atom.setX(xString); atom.setY(yString); atom.setZ(zString);
atom.setId(id);
if (line.substring(0, 6).trim().equals("ATOM"))
atom.setStandard("true");
else atom.setStandard("false");
// Atom speichern, um spaeter die Bindungen zu berechnen
// und auf die Daten zugreifen zu koennen
residueBox.storeAtom(atom); atomTable.put(atomId.trim(),atom);
}
|
public void conect(String line) {
// ueberprueft, ob die Flagge -c gesetzt wurde
// wenn nein wird die Zeile ignoriert
if (conect.booleanValue()) {
// auslesen der Daten
String mainAtom = line.substring(6, 11).trim();
String bondedAtom_1 = line.substring(11, 16).trim();
String bondedAtom_2 = line.substring(16, 21).trim();
String bondedAtom_3 = line.substring(21, 26).trim();
String bondedAtom_4 = line.substring(26, 31).trim();
// versucht entsprechende Bindung anzulegen
createBond(mainAtom, bondedAtom_1);
createBond(mainAtom, bondedAtom_2);
createBond(mainAtom, bondedAtom_3);
createBond(mainAtom, bondedAtom_4);
}
}
|
public void model(String line) {
// Nummer des Modells
String modelCounter = line.substring(10, 14).trim();
// ist Nummer > 1 werden neue Knoten
// in den DOM-Tree eingehangen
if (!modelCounter.equals("1")) {
Element atomArray = document.createElement("atomArray");
atomArray.setAttribute("id", "Model " + modelCounter);
// ermittelt passende Position des neuen Knotens
// und haengt ihn ein
Element moleculeNode = (Element) document.getLastChild();
moleculeNode.appendChild(atomArray);
// loescht bisherige Atomdaten, da Bindungsverhaeltnisse
// aller Modelle gleich sind
residueBox.clear();
}
}
|
public void end(String line) {
// startet die Berechnung der Bindungen
Vector bondBox = residueBox.makeBonds(conect);
// bondArray in den DOM-Tree einfuegen
Element moleculeNode = (Element) document.getLastChild();
Element bondArray = document.createElement("bondArray");
moleculeNode.appendChild(bondArray);
for (int i = 0; i < bondBox.size(); i++) {
Bond bond = (Bond) bondBox.elementAt(i);
createBond(bond);
}
}
|