

Nächste Seite: Resumée und Ausblick
Aufwärts: Diplomarbeit
Vorherige Seite: Implementierung
Inhalt
Anwendung
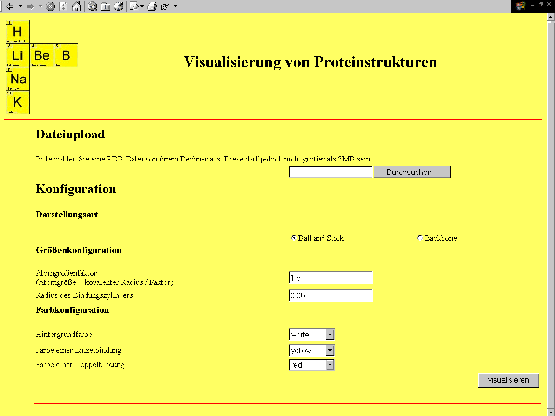
Als konkrete Anwendung wurde eine Webapplikation erstellt, mit der ein Nutzer eine PDB-Datei von seinem Dateisystem auf den Server laden und sich nach der Konvertierung direkt in einer 3D-Szene anzeigen lassen kann. Dabei stehen zwei Auswahlmöglichkeiten zur Verfügung, der Standard-Modus und der Experten-Modus. Über eine Oberfläche kann der Benutzer Einfluß auf die endgültige Darstellung nehmen. Im Standard-Modus lassen sich folgende Werte konfigurieren:
- der Atomgrößenfaktor
- der Radius des Bindungszylinders
- die Farbe des Hintergrunds
- die Farbe einer Einfachbindung
- die Farbe einer Doppelbindung
Die zugehörige Oberfläche ist in Abbildung 8.1 zu sehen.
Abbildung 8.1:
Oberfläche der Webapplikation
|
|
Im Experten-Modus können zusätzlich die einzelnen Atome konfiguriert werden. Zum Einen kann die Farbe jedes Atoms festgelegt werden und zum Anderen lassen sich die kovalenten Radien der Atome angeben. Da diese eine Grundlage bei der Berechnung der Bindungen bilden und zu einem Atom unterschiedliche Werte zu finden sind, wird hier dem Benutzer die Gelegenheit gegeben, eigene Werte zu verwenden. Wird z.B. der kovalente Radius eines Elements auf 0 gesetzt, werden keine Bindungen zu diesem Element generiert und dementsprechend nicht in der Szene angezeigt.
Die voreingestellten Werte werden aus den Konfigurationsdateien gelesen und in den entsprechenden Fomularfeldern angezeigt. Die eingegebenen Daten werden in neu angelegten Dateien geschrieben, die bei den Konvertierungen in den jeweiligen Kommandoaufrufen mit angegeben werden.
Als Beispiel wurde das Protein Insulin visualisiert. Mit den voreingestellten Werten ergibt sich eine Darstellung, ähnlich wie Ball and Stick. Besonders zu beachten sind die unterschiedlichen Farbwerte der verschiedenen Bindungen.
Abbildung 8.2:
Ball and Stick-Darstellung des Insulin
|
|
Wird der Atomgrößenfaktor auf 0.8 geändert, wird eine Darstellung, die Spacefill ähnelt, erzeugt. Hierbei sind die Bindungen untereinander nicht mehr zu sehen.
Abbildung 8.3:
Spacefill-Darstellung des Insulin
|
|
Wird die Darstellungsart Backbone gewählt und wird der Atomgrößenfaktor auf 2,5, die Hintergrundfarbe auf schwarz und die Farbe einer Einfachbindung und die der Kohlenstoffatome auf weiß gesetzt, wird folgende Darstellung generiert:
Abbildung 8.4:
Backbone-Darstellung des Insulin
|
|
Besonders gut sichtbar sind die verschiedenen Bindungen mit ihren unterschiedlichen Farbwerten in der Darstellungsart Wireframe. Diese wird erzeugt wenn der Atomgrößenfaktor z.B. auf 100 gesetzt wird. Damit sind die Atome nicht mehr sichtbar.
Abbildung 8.5:
Wireframe-Darstellung des Insulin
|
|



Nächste Seite: Resumée und Ausblick
Aufwärts: Diplomarbeit
Vorherige Seite: Implementierung
Inhalt
Oliver Krone
2003-04-28